■ 서포트 벡터 머신(330p)
3차원을 초평면 기법으로 분류하는 머신러닝
아래의 검정돌과 하얀돌을 잘 분리하고 있는 선이 어디 입니까?
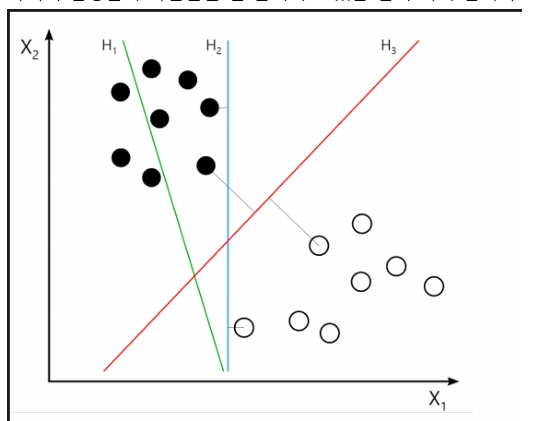
답: 빨간색선
아래에서 좀 더 여유있게 분류를 하고 있는 선은 B1 입니까?

답: B1 이 좀 더 여유있게 분류를 하고 있다.
이 둘 사이의 거리를 마진(margin) 이라고 한다.
이 선은 단순히 생긴 선이 아니고, 수학적으로 계산이 되어서
이루어진 선이다. 이 선을 분리선이라고 부르고 초평면(hyperplane)
이라고 부르기도 한다.
위에서 분리를 하고 있는 그림은 2차원 그림이고 3차원 그림으로
다시 나타내면 아래와 같다.
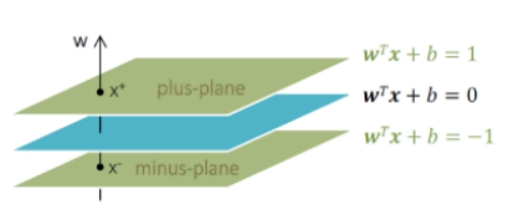
평면 사이의 경계가 margin 이다.
수직으로 관통하는 선 w 를 벡터(방향+힘) 라고 한다.
W : 가중치
b : 바이어스
X : 입력 데이터
WT*X + b = 1 ***개고양이 할 때 맞추면 1 , 틀리면 0 이였던것과 같음
WT*X + b = -1 *** 틀리면 SVM은 -1 이다.
행렬의 전치는 대각선을 변경하는 건데
벡터의 전치는 방향이 90도로 변경된다.

여기서 여유있게 분리하는 마진 선에 걸쳐있는 데이터를
서포트벡터 라고 한다.
333p
서포트벡터는 각 클래스에서 MMH에 가장 가까운 점들이다.
서포트 벡터만을 사용해서 MMH를 정의할 수 있다.
*MMH: 최대마진초평면(Maximum Margin Hyperplane)


이 과정의 목표는 이 공식을 이용해 두 초평면을 명시하는
가중치의 집합을 찾는 것이다
두 평면간의 거리를 계산을 해야한다
만약에 2차원에서는 점과 점사이의 거리를 구하면 되지만
3차원에서는 평면과 평면 사이의 거리를 구해야 해서
벡터로 (평면과 평면의 경계를 위한) 거리를 구해야 한다.

두 평면간의 거리를 다음과 같이 정의한다
||w|| : 유클리드놈 (원점에서 벡터w 까지의 거리)
2
--------- : 두평면간의 거리를 정의한다.
||w||
이 값이 margin 이고 최대값이 되는걸 목표로 한다.

두 평면사이의 마진을 최대화 하는 공식 max 에 있는
분자 분모를 뒤집어서 min 에 나타낸 식이다
위의 가중치 벡터인 W를 알아내는 것이 서포터머신 알고리즘 훈련의
목표이다.
■ 서포트 벡터 머신에서 오버피팅을 줄이는 방법

C가 추가되면서 W 벡터에 대한 패널티를 부여하고 있다.
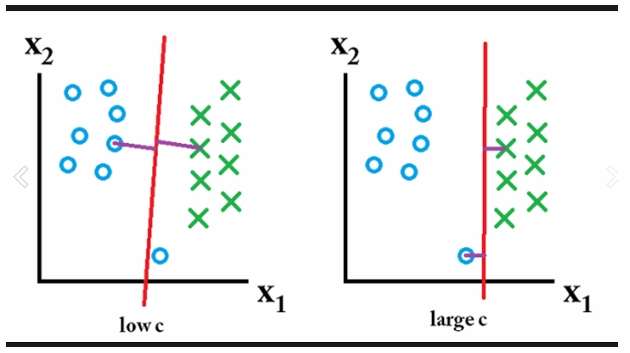
C값을 변경해 가면서 오버피팅을 예방할 수 있다.
337p
비용파라미터 C.
예를 들어 초평면의 잘못된 쪽에 데이터 포인트가 있다면
C 값을 변경해서 패널티를 조정할 것이다.
비용 파라미터가 커질수록 최적화 알고리즘이 100% 분리를 해내는 것이
더욱 어려워질 것이다.
한편 비용 파라미터가 작아지면 전반적으로 넓어진 마진을 중요시할 것이다.
잘 된 모델을 만들려면 둘 사이의 균형을 맞추는 것이 중요하다.
C값을 낮게 잡아야 오버피팅을 낮출 수 있다.
C값을 높게 잡으면 정확도는 올라가지만 마진값이 줄어든다.
실습1. 필기체 데이터 SVM 으로 분류 모델 생성하기(알파벳)
|1. 데이터 로드
letters <- read.csv("letterdata.csv")
str(letters)
2. 훈련 데이터와 테스터 데이터 구분 ( 8대 2로 나눈다)
letters_train <- letters[1:16000, ]
letters_test <- letters[16001:20000, ]
3. 데이터로 모델 훈련 ----
# 단순 선형 SVM을 훈련으로 시작
install.packages(“kernlab”)
library(kernlab)
letter_classifier <- ksvm(letter ~ ., data = letters_train, kernel = "vanilladot")
#개,고양이는 단 2개라서 선으로 분류가 가능했지만
# 알파벳같이 26개의 분류대상은 비선형으로 처리해야 한다.
# vanilladot (바닐라아이스크림 땡땡이)
4. 모델에 대한 기본 정보 확인
letter_classifier
5..모델 성능 평가 ----
# 테스트 데이터셋에 대한 예측
letter_predictions <- predict(letter_classifier, letters_test)
head(letter_predictions)
table(letter_predictions, letters_test$letter)
6. 일치/불일치 예측을 표시하는 TRUE/FALSE 벡터 생성
agreement <- letter_predictions == letters_test$letter
table(agreement)
prop.table(table(agreement))
agreement
FALSE TRUE
0.16075 0.83925
문제277.
위의 필기체를 구분하는 svm 모델의 정확도를 올리기 위해서
ksvm 함수의 kernel 파라미터값을 rbfdot 로 변경해서 수행하시오
3. 데이터로 모델 훈련 ----
install.packages(“kernlab”)
library(kernlab)
letter_classifier <- ksvm(letter ~ ., data = letters_train, kernel = "rbfdot")
prop.table(table(agreement))
agreement
FALSE TRUE
0.07025 0.92975
문제278.
한국인 신체지수 데이터를 이용해서 복부비만인지 정상인지를
예측하는 svm 모델을 생성하시오
# csv 파일 불러옴(이전에 안에 있는 한글 전부 영어로 다 바꿈)
body <- read.csv("kbody2.csv", header = F)
head(body) # V20 이 라벨이다.
# 라이브러리 불러옴
library(e1071)
# na값 제거
body1 <- na.omit(body)
# 컬럼명 줌
colnames(body1) <- c("gender", "age", "height", "chest", "heory", "bae", "ass", "kyeo",
"face_vertical", "head", "bone", "body_fat", "body_water",
"protein", "mineral", "body_fat_per", "bae_fat_per", "work", "bae_fat_test",
"work_test")
컬럼 설명 : 성별 나이 키 가슴둘레 허리둘레 배둘레 엉덩이둘레
겨드랑둘레 얼굴수직길이 머리둘레 골격근량 체지방량 체수분
단백질 무기질 체지방률 복부지방률 기초대사량 복부지방률평가
대사량평가
# 랜덤시드 생성
set.seed(12345)
# 셔플
body_ran <- body1[order(runif(12894)), ]
# 트레이닝셋 80%
body_train <- body_ran[1:10314, ]
# 테스트셋 20%
body_test <- body_ran[10315:12894, ]
# 선형SVM 훈련
body_svm <- svm(work_test~., data = body_train, kernel="linear")
body_svm
# 모델 테스트
p <- predict(body_svm, body_test, type="class")
p
table(p, body_test[, 20])
# 분류 결과 확인
mean(p == body_test[, 20])
0.9418605
문제279.
한국인 신체지수 데이터의 svm 모델의 정확도를 올리시오
?svm
body_svm <- svm(work_test~., data=body_train, kernel='radial')
mean(p == body_test[, 20])
[1] 0.9550388
'R' 카테고리의 다른 글
| 특화된 머신러닝 주제 (0) | 2019.04.17 |
|---|---|
| 랜덤포레스트 (0) | 2019.04.17 |
| 모델성능개선 부스팅, 앙상블,배깅 (0) | 2019.04.17 |
| 모델 성능개선 (0) | 2019.04.03 |
| 모델 성능평가 (0) | 2019.04.03 |


